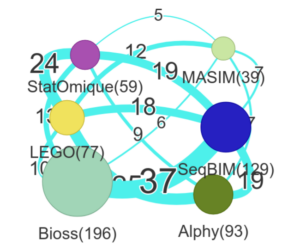
Le GDR BIMMM anime 6 groupes de travail thématiques (communs pour certains avec d’autres GDR) qui proposent une animation scientifique ciblée tout au long de l’année. Ce sont des lieux d’échanges et de discussions dont l’action s’inscrit dans la durée. Ils proposent des rendez-vous réguliers (au moins une fois par an en présentiel et pour certains de façon régulière à distance) et conviviaux. L’inscription se fait via la plateforme myGDR (procédure détaillée en fin de page).
AlPhy Alignement et Phylogénie
ALPHY, crée en 1995, est dédié à la génomique évolutive : ALPHY = ALignement + PHYlogénie. Alphy permet depuis près de 30 ans à la communauté de se rencontrer et de présenter ses travaux dans le domaine de l’évolution moléculaire, de la modélisation des séquences, de la génomique des populations, de la reconstruction de l’histoire évolutive du vivant, etc. L’explosion des techniques de séquençage, des capacités de calculs et l’accumulation de données a permis des avancées spectaculaires et des changements d’échelle dans notre compréhension de l’organisation, du fonctionnement et de l’évolution des génomes, et dans nos connaissances sur l’histoire évolutive du vivant, à toutes les échelles de temps. La génomique évolutive, initialement focalisée sur l’étude des divergences entre espèces (phylogénomique), s’est ainsi progressivement étendue à l’analyse de la diversité intra-spécifique (génomique des populations). Ces avancées reposent sur des développements bioinformatiques pour tirer le meilleur parti des progrès des techniques de séquençage ainsi que sur des développements théoriques en évolution moléculaire et génétique des populations.
L’ambition d’ALPHY, depuis son origine, est de favoriser les échanges informels dans un champ fortement interdisciplinaire, et, notamment, de permettre aux jeunes chercheurs du domaine de s’exprimer. Chaque année, ALPHY organise ses journées annuelles, qui regroupent entre 100 et 150 participants sur 2 ou 3 jours.
BIOSS Biologie Systémique Symbolique
BIOSS, commun avec le Groupe de recherche Informatique Mathématique (GDR IM), a pour thème la BIOlogie Systémique Symbolique, avec les méthodes et outils autour de la modélisation des systèmes biologiques. Les intérêts de BIOSS sont à la frontière de l’informatique fondamentale, des mathématiques discrètes et de la biologie moléculaire, avec des applications liées à la santé.
Les contributions incluent des travaux liés à une meilleure compréhension du fonctionnement des systèmes biologiques en permettant de les étudier et de les manipuler in silico. L’objectif est de prédire des comportements cellulaires tout en prenant en compte les multiples échelles spatiales et temporelles qui dépendent des interactions entre gènes, protéines, métabolites, cellules ou populations. Parmi les principales questions on peut citer : En quoi les systèmes biologiques sont-ils formalisables ? Quels nouveaux mécanismes de transmission d’information sont-ils à l’œuvre en biologie moléculaire et cellulaire ? Mais également, qu’est-ce que l’informatique peut apporter à la biologie, au-delà des approches de modélisation numérique ?
BIOSS comprend plus de 200 participants venant de plus de 40 équipes différentes. La communauté BIOSS se réunit via des séminaires virtuels mensuels, chaque premier vendredi du mois, et deux journées annuelles : la journée du GT et une journée thématique
SeqBIM Séquences en Bioinformatique, Informatique et Mathématiques
SeqBIM, commun avec le Groupe de recherche Informatique Mathématique (GDR IM), traite de l’algorithmique des séquences, de toutes les séquences, des plus courtes aux plus longues, qu’elles soient d’ADN, d’ARN, ou de protéines. Historiquement, la bioinformatique est très liée à l’algorithmique des séquences. On peut par exemple penser à l’algorithme d’alignement Needleman et Wunsch publié il y a plus de 50 ans ! Depuis, ce lien n’a pas faibli et les questions sont sans cesse renouvelées : assemblage de génomes, de transcriptomes, de métagénomes, alignement, analyse de pangénomes, traitement des longues lectures… Il y a derrière des problématiques difficiles d’algorithmique discrète et d’indexation de données sur les mots, les arbres, les graphes. La communauté est très active pour proposer des solutions capables d’absorber les débits toujours plus importants des séquenceurs à haut-débit.
Le groupe rassemble actuellement environ 150 scientifiques travaillant sur les méthodes pour traiter et analyser des séquences biologiques. Il se structure principalement autour de ses journées annuelles, qui réunissent 50 à 70 personnes pendant deux jours et qui sont l’occasion de présenter les derniers travaux sur les thématiques du groupe. En particulier, c’est une excellente opportunité pour les jeunes chercheurs et chercheuses d’intégrer la communauté et d’y présenter leurs premiers résultats.
LEGO Machine Learning pour la Génomique
LEGO, crée en 2023, est dédié au machine learning pour la génomique. Comme de nombreux domaines scientifiques, la génomique bénéficie de la montée en puissance qu’a connu le machine learning au cours de la dernière décennie. La communauté française de recherche en machine learning pour la génomique est active mais dispersée sur le territoire au sein d’équipes traditionnellement expertes dans d’autres domaines méthodologiques pour la génomique tels quel la statistique, l’algorithmique ou la bioinformatique. L’objet du groupe de travail LEGO est de faciliter les échanges au sein de cette communauté, de favoriser les collaborations, et de permettre le suivi des progrès effectués par les équipes participantes.
Le groupe se réunit lors de journées annuelles et d’autres manifestations adossées par exemple aux journées du GDR ou à JOBIM.
MASIM Structures et Interactions Macromoléculaires
MASIM, créé à l’Automne 2017, est dédié à la bioinformatique structurale, et plus particulièrement aux aspects méthodologiques sous-jacents aux approches bioinformatiques en biologie structurale. Il vise à susciter de nouvelles synergies au sein d’une communauté traditionnellement morcelée par la diversité des méthodes, niveaux de granularité et types de macromolécules d’intérêt.
Le groupe se réunit lors de journées annuelles et contribues à l’organisation de rencontres thématiques et d’écoles-chercheurs.
StatOmique Modélisation et analyse statistique de données omiques
Statomique, créé en 2008, est basé sur le partage d’expérience autour de la modélisation et l’analyse statistique de données omiques. Le groupe compte aujourd’hui plus de 100 abonnés d’horizons divers (académique, privé), travaillant aux quatre coins de la France et même parfois plus loin. Il rassemble une communauté de statisticiens et bioinformaticiens, recouvrant des profils variés, allant de la conception de la méthode, au développement de l’outil jusqu’à l’analyse et l’interprétation des données.
Le groupe se réunit lors de journées ou demi-journées à thème (2 à 4 par an) avec comme ligne directrice : le partage d’expériences sur la modélisation et l’analyse statistique de données.
Inscription aux GT du GdR
L’inscription aux GT se fait via la plateforme myGDR en suivant la procédure suivante
- Allez sur https://mygdr.hosted.lip6.fr/
- Cliquez sur « Laboratoires » afin de vérifier que votre laboratoire est présent dans la base. (Si votre laboratoire n’est pas présent, vous ne pouvez pas vous inscrire dans l’immédiat. Envoyez-nous par mail gdr-bim@services.cnrs.fr : le nom complet de votre laboratoire, son acronyme, son adresse web.)
- Cliquez sur « S’inscrire », et procédez à l’inscription (e-mail professionnel de préférence) en respectant les consignes de création de mot de passe, et en mémorisant vos login et mot de passe.
- Connectez-vous avec votre nom de login de votre mot de passe.
- Remplissez votre fiche.
- Cliquez sur « BIM », et la liste des GT apparaît. Sélectionnez le ou les GTs au(x)quel(s) vous souhaitez appartenir.
Vous êtes déjà plus de 400, issus de plus de 140 laboratoires distincts à l’avoir fait avec des recoupements importants entre GT.